Projet PEHAGA
Les plates-formes informatiques hétérogènes (HCP) sont des systèmes informatiques constitués de combinaisons de différents types d'éléments de traitement. Le type d'élément de traitement emblématique de l'unité centrale de traitement (CPU) est utilisé pour la grande majorité des calculs par les ordinateurs dans le monde, mais l'intérêt pour les HCP augmente. Cette croissance est liée à l'accessibilité à de nouveaux outils pour la programmation de ces éléments hétérogènes.
Les unités de traitement graphique (GPU), qui sont l'un de ces types d'éléments, sont devenues plus accessibles aux programmeurs pour des tâches qui ne sont plus exclusivement liées aux graphiques. De même, les FPGAs, qui sont des circuits permettant de «programmer» le matériel, sont des dispositifs programmables qui peuvent être reconfigurés à la volée comme si l’on créait un nouveau circuit électronique à chaque fois.
Les FPGA sont utilisés depuis des années dans les routeurs et les marchés de niche, mais, à l’instar des GPU, ils sont devenus plus accessibles aux programmeurs pour d'autres tâches informatiques. Une des conséquences de cette accessibilité accrue est que les HCP sont devenus stratégiquement importants pour les centres de données, qui peuvent utiliser des combinaisons de différents types d'éléments de traitement pour une plus grande efficacité de calcul et des économies d'énergie. L'acquisition récente par Intel Corporation de l'un des deux plus grands fabricants de FPGA, Altera Corporation, a clairement marqué cette tendance vers les HCP. Intel a ensuite annoncé la disponibilité prochaine d'un HCP composé d'un processeur Intel avec un FPGA Altera dans le même package.
Cette tendance à l’exploitation de systèmes hétérogènes est mondiale. Les centres de données et de traitement des géants de l’informatique (GAFAM) ont annoncé leur intention d'utiliser des HCP avec des FPGA pour économiser de l'énergie. Cependant, la conception de systèmes avec FPGA n'est pas simple, car des compétences spéciales sont requises, rarement possédées par les développeurs de logiciels traditionnels.
Un domaine dans lequel les HPC seront clairement un plus est celui de la génomique. Plus précisément, le domaine s'intéresse à une série d'étapes allant des données brutes aux génomes entièrement analysés que nous appelons ici le Génome Processing Pipeline (GPP). La demande d'amélioration du GPP est à la hausse. Avec l'avènement du séquençage à haut débit, la génomique est entrée dans une nouvelle ère où des quantités massives de données sont produites (2-40 ExaBytes/an en 2025 sont attendus). Le séquençage d'un génome humain génère de l'ordre de 100 Go de données brutes compressées. Ces données sont de petites séquences (également appelées «reads»), localisées au hasard dans le génome, avec une redondance élevée (généralement 30x-50x). Même lorsqu'il se concentre uniquement sur l'exome (la partie codante de l’ADN chromosomale), le séquençage génère toujours 10 Go de données brutes par génome. Le traitement de ces données en un temps limité est actuellement l'étape limitative du processus de séquençage du génome. Par exemple, il faut environ 700 minutes pour traiter un seul ensemble de données de séquençage d'exome sur un processeur AMD standard à 8 cœurs et environ 7 jours sur un ordinateur 32 cœurs pour un génome entier.
Si l'accélération fait partie de l'objectif de ce projet, un effet très important de l'exploitation des FPGA réside dans la réduction de la consommation d'énergie. Ils peuvent consommer beaucoup moins d'énergie que les processeurs standard pour le même calcul (par exemple, Microsoft l'a réduite de 50% pour son moteur de classement de pages Bing). Dans un monde où l’économie d’énergie devrait guider nos développements afin de créer des systèmes «durables», la réalisation des objectifs de ce projet réduira considérablement les coûts de fonctionnement des applications d'analyse du génome à grande échelle et permettra à une grande variété d'hôpitaux et d'institutions d'acquérir et d'exécuter des analyses de données massives.
A l’heure actuelle, le projet a vu la réalisation de deux systèmes, tous deux publiés sous forme d’articles scientifiques :
1. Réalisation d’un système de comptage de K-mer sur FPGA. Les K-mer sont des séquences d’ADN d’une certaine longueur, et le but de l’application était de compter combien chacune de ces séquences est présente dans les éléments séquencés. Cet algorithme permet ensuite d’aider la reconstruction du génome à partir de tous les brins observés. Ceci a été réalisé sur une carte pocédant 6 FPGAs et 6 mémoires HMCs. Ces mémoires séries hyper performantes offrent la possibilité de réaliser des opérations arithmétiques directement dans la mémoire, sans transfert de données. Ce nouveau type d’architecture nous a permis d’améliorer les performances du système développé et d’exploiter pour la première fois ce type de calcul.
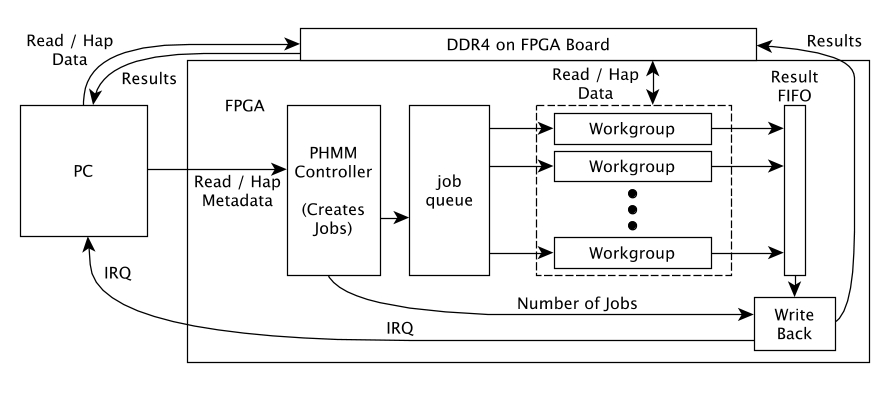
2. Réalisation de l’algorithme pair-HMM sur FPGA. Cet algorithme est au cœur d’un des pipelines de traitement traditionnel, et représente 20-25 % du temps de traitement du logiciel. Il implique des calculs avec nombres réels, et un passage au travers d’une matrice (typiquement fait en programmation dynamique), afin de calculer la vraisemblance d’alignement de deux séquences génomiques. La réalisation a été intégrée dans le cloud Amazon. Amazon offre en effet la possibilité d’utiliser des cartes FPGA (appelées F1) dans le cloud. Le logiciel embarquant l’accélérateur FPGA a donc été exécuté dans le cloud. Ceci était une première pour l’institut REDS, et permet de disposer d’un système potentiellement clonable pour d’autres utilisateurs, sans besoin d’acheter du matériel spécifique.


